Definición de ADN Mitocondrial

Lic. en Antropología Física
El genoma mitocondrial es una molécula circular de doble hélice, cerrada y pequeña, de 16 569 pares de bases (pb) que se hereda sólo de madres a hijos e hijas (son raros los casos de heteroplasmia), por lo que no hay recombinación genética al momento de la concepción, además evoluciona exclusivamente por acumulación de mutaciones en el tiempo.
La alta tasa de mutación del genoma mitocondrial (de 10 a 20 veces que el ADN nuclear, en cuanto a genes de funciones comparables) resulta de utilidad para diferenciar entre poblaciones que a lo largo del tiempo han estado relacionadas biológicamente, sin embargo, su tasa de mutación puede ser tan rápida que pueden ocurrir fenómenos de retromutación. La tasa de evolución media de esta molécula ha sido calculada a partir de especies en las que se disponía de tiempos de divergencia de los restos fósiles, y de datos biodemográficos o proteicos; el resultado da 1-2% por millón de años, válido para diferentes órdenes.
Este ADN particular funciona como herramienta para investigar el parentesco biológico entre poblaciones debido a que sus propiedades permiten dilucidar las relaciones entre poblaciones que han divergido en tiempo reciente, sin tener que considerar los fenómenos de recombinación y añadiendo una dimensión temporal; para ello se debe tener en mente lo siguiente: dos individuos cuyo ancestro común haya sido mujer, tendrán moléculas de ADN mitocondrial tan diferentes como tiempo haya transcurrido desde la separación del ancestro.
Haplogrupos para la clasificación del ADN mitocondrial en la distribución geográfica
Algunas regiones de ADN pueden resultan muy similares entre sí, permitiendo su clasificación en un mismo conglomerado, lo cual se conoce como haplogrupo. Por ejemplo, Torroni et al (1993) tipificaron los linajes fundadores mitocondriales para el Continente Americano, los cuales recibieron la denominación de haplogrupos A, B, C, y D, de acuerdo a mutaciones puntuales de la secuencia mitocondrial. Estas mutaciones crean diferentes sitios de corte para enzimas específicas, como se describe a continuación: en el linaje A existe el sitio de corte 663 para la enzima HaeIII, el linaje C se caracteriza por el sitio 13259 para HincII, en el linaje D se reconoce el sitio 5176 para AluI; esta identificación se realiza a partir de polimorfismos de fragmentos de longitud (RFLP). Para el caso del linaje B, existe una deleción de 9 pares de bases en la posición 8272-8289.
Se ha descrito la distribución geográfica de cada linaje de la siguiente manera: el linaje A es el predominante en el Continente Americano y sobre todo en Norteamérica; sin embargo, este haplogrupo también es atribuido a poblaciones mesoamericanas. Los linajes C y D aparecen sobre todo en Sudamérica; el linaje B se encuentra en la región norte y sur de la costa del Pacífico, además Kemp et al (2010) lo propone como un linaje característico de la familia yuto-azteca y del suroeste de E.U.A.
Además, existen mutaciones que se comparten entre individuos de un mismo haplogrupo, lo que ha permitido describir haplotipos (o sublinajes) específicos de algunas poblaciones. En la Figura 1 se pueden observar las regiones del ADN mitocondrial que contienen las mutaciones para reconocer cada linaje.
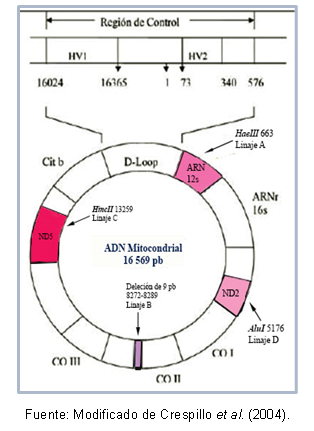
Figura 1. Regiones del ADN mitocondrial que tipifican cada linaje fundador americano.
Durante algunos años sólo se reconocieron estos cuatro linajes hasta que se incluyó el haplogrupo X para las poblaciones del norte del continente; el cual también está lejanamente relacionado con poblaciones europeas. Los linajes anteriores tienen una correspondencia con poblaciones asiáticas, aunque se observan en menor frecuencia que en el Continente Americano; los linajes A, B y C no se encuentran en africanos y caucásicos actuales; y el linaje D también existe en África, pero asociado a otros sitios de restricción. Con lo anterior podríamos decir que estos haplogrupos caracterizan a las poblaciones americanas y por lo tanto su estudio es adecuado en cuanto a poblamiento y migraciones (antiguas y recientes).
Distancias genéticas
La manera de establecer similitudes o diferencias genéticas entre poblaciones, es con las distancias genéticas, las cuales pueden tener una explicación histórica, dado que cambian (aumentan o disminuyen) al cabo del paso de las generaciones, y pueden dirigirnos a sucesos en la historia de una población por ejemplo: al momento de una gran migración o al contacto entre dos culturas, con este cálculo podemos discernir qué mecanismos entraron en acción para darnos tal o cual resultado.
Tanto la deriva genética como el flujo génico tienen que ver con la facilidad en que un grupo, en este caso de seres humanos, tiene para moverse dentro de un territorio y de entrar en contacto con otros grupos. Entonces, el aislamiento geográfico es la distancia genética que crece con el aumento de la distancia geográfica entre los grupos humanos.
Hipótesis sobre la diversidad del ADN mitocondrial
Para explicar la diversidad en los linajes mitocondriales fundadores en América, existen dos hipótesis: que este continente haya sido colonizado por múltiples eventos desde Beringia, o que ya ocurrida la migración hayan ocurrido cambios evolutivos después de la colonización. También se explican dos rutas de entrada de estas variaciones al continente, la primera propone que los cuatro haplogrupos fundadores sin variaciones, es decir, cada uno con un haplotipo raíz, pudieron llegar justo después del Último Máximo Glaciar o un poco antes, con fechas de hace 21 mil a 19 mil años y habrían seguido una ruta costera por el Pacífico; la segunda propuesta sugiere que estas variaciones intra-haplogrupo ya existían en Beringia y fueron llevadas hacia el sur del continente Americano pero su entrada habría sido exactamente al final del Último Máximo Glaciar de manera que las rutas dentro del continente ya estarían libres, entonces la entrada de estos grupos humanos sería hace 19 mil años. Se encuentra además una gran diversidad del haplogrupo A y un tiempo de coalescencia para éste que es menor que el resto (17 mil años).
La explicación más probable para esto es que se deba a expansiones secundarias del haplogrupo A desde Beringia, mucho después del final del Último Máximo Glaciar. A pesar de la discrepancia en cuanto al tiempo de entrada del hombre en América, los estudios genéticos han logrado dar cierta claridad, porque apoyan la hipótesis de que existían grupos humanos en el continente americano antes de clovis; y se encuentra que hay una separación entre los ancestros del noreste asiático hace 25-35 mil años y la entrada en América hace 15-35 mil años. En la Figura 2 se observan las rutas de los haplogrupos mitocondriales en el mundo y el tiempo de divergencia en años antes del presente.
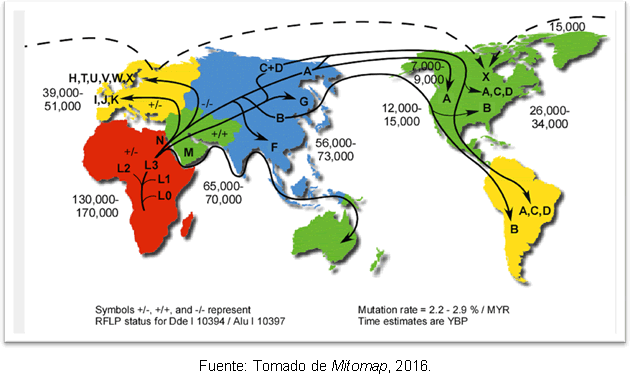
Figura 2. Mapa de las diferentes rutas de dispersión de los linajes mitocondriales
Importancia de la filogeografía en el estudio genético y los datos de los genes mitocondriales
Existe una herramienta de gran utilidad para los análisis genéticos; la filogeografía. Se trata de las primeras aplicaciones de los estudios moleculares, y con ella se busca poder determinar las relaciones filogenéticas y espaciales entre secuencias de nucleótidos, en este caso de ADN mitocondrial. La distribución espacial puede asemejar un patrón temporal, es decir, las secuencias de ADN más distantes geográficamente deberían ser las más diferentes genéticamente, así como las secuencias de ADN que divergieron hace mucho tiempo también deberían ser las más diferentes genéticamente. Entonces las poblaciones distantes geográficamente, con muy poco o nada de flujo génico entre ellas, acumularían diferencias debido a la deriva génica y la mutación, incluso por selección; pero pueden ocurrir situaciones que no permitan la deriva, como uno o varios efectos fundadores u otros patrones de flujo génico.
Los datos que proveen los genes mitocondriales han sido de los más útiles en las investigaciones de filogeografía por la característica que estos genes tienen de no recombinarse y como resultado muestran una línea filogenética mucho más clara que muchos genes nucleares. El tamaño efectivo de población calculado con los genes mitocondriales (y con el cromosoma Y que funciona de una manera similar) es aproximadamente un cuarto del calculado para los genes nucleares, entonces la divergencia ocurre casi cuatro veces más rápido que con los genes nucleares, este ritmo rápido de divergencia (y de flujo génico) puede causar que el patrón observado de herencia en estos genes uniparentales sea diferente que las filogenias obtenidas con genes nucleares (los cuales representan la mayoría del acervo genético en un individuo).

Trabajo publicado en: Jul., 2022.
Referencias
Achilli, A., Perego, U. A., Bravi, C. M., Coble, M. D., Kong, Q.-P., & et al. (2008). The Phylogeny of the four Pan-American MtDNA Haplogroups: Implications for Evolutionary and Disease Studies. PLoS ONE , 3 (3), e1764.Cavalli-Sforza, L. L. (2000). Genes, pueblos y lenguas (1ª Edición en Biblioteca de Bolsillo ed.). Barcelona, España: Editorial Crítica.
Rebato, E., Susanne, C., & Chiarelli, B. (Edits.). (2005). Para comprender la Antropología Biológica: evolución y biología humana. Verbo Divino.
Torroni, A., Schurr, T. G., Cabell, M. F., Brown, M. D., Neel, J. V., Larsen, M., y otros. (1993). Asian affinities and continental radiation of the four founding Native American mtDNAs. The American Journal of Human Genetics , 53 (3), 563.
(2022). ADN mitocondrial, National Human Genome Research Institute.
-Referencia del esquema del ADN mitocondrial-
Crespillo, M., Paredes, M., Arimany, J., Guerrero, L., & Valverde, J. (2004). Guerra Civil Española (1936-1939): identificación de restos humanos procedentes de fosas comunes en Cataluña mediante análisis de ADN mitocondrial. A propósito de un caso. Cuadernos de Medicina Forense (38), 37-46.
Escriba un comentario
Contribuya con su comentario para sumar valor, corregir o debatir el tema.Privacidad: a) sus datos no se compartirán con nadie; b) su email no será publicado; c) para evitar malos usos, todos los mensajes son moderados.